はじめに
target trial emulationについてまとめた中で不死時間バイアス:Immortal time biasを知りました。
Rのコードは以下の記事をかなりパクる参考にさせてもらっている。
moratoriamuo.hatenablog.com
moratoriamuo.hatenablog.com
不死時間バイアス
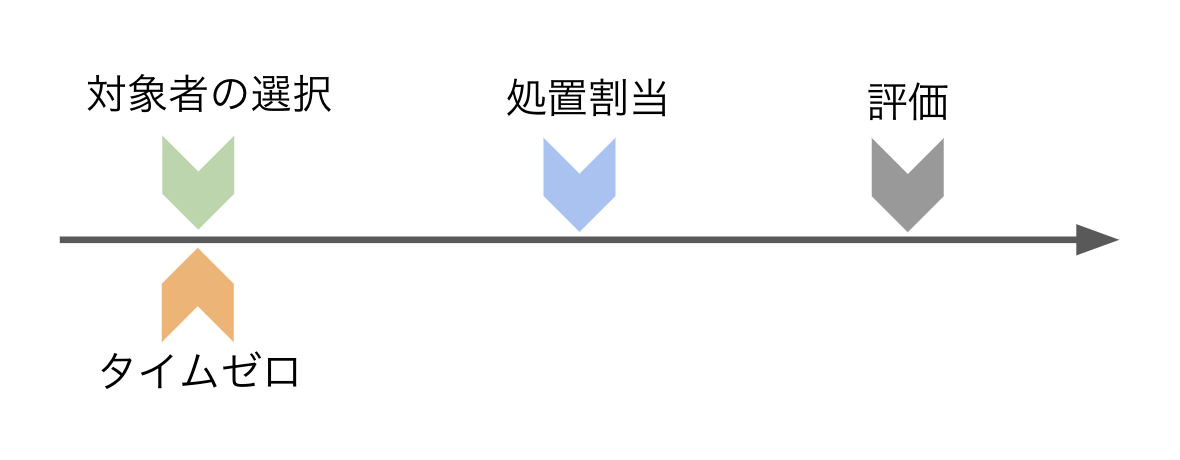
フォローアップ開始から治療開始までに時間がある場合、その期間はイベントが起きない。
もし、イベントのが治療開始までにイベントが発生してしまった場合は、対照群に割り当てられることになる。
この時、フォローまでは絶対にイベントが起きないため、その分生存時間が伸びることになる。
このバイアスを不死時間バイアス:Immortal time biasと呼ぶ。
www.bmj.com
RでImmortal time biasの確認
RでImmortal time biasが本当に発生するのかを確認する。
生存時間情報はワイブル分布を用いて生成する。
処置群のうち確率的に生成されたタイミングで処置が行われるとする。
この時、処置タイミングより前に生存時間に達してしまっている場合は、対象群に割り当てる。
一回1000サンプルを生成し、1000回繰り返すことで、バイアスの状況を分布として確認する。
library(tidyverse)
library(survival)
library(survminer)
library("progress")
gamma = 1.0
N = 1000
L = 1000
result_df <- data.frame()
pb <- progress_bar$new(total = L)
for(l in 1:L){
pb$tick()
suvival_df <- data.frame(id = 1:N,
Lambda = 1) %>%
mutate(time1 = rweibull(N, shape=1.0, scale=Lambda),
time2 = rweibull(N, shape=1.0, scale=Lambda*exp(-log(gamma))),
treat = rbernoulli(N, p=0.5),
treat_time = rweibull(N, shape=1.0, scale=0.6*Lambda),
cons_time = runif(N, 0, 5)
) %>%
mutate(time = if_else(treat==1, time2, time1),
treat = if_else((time2 < treat_time)&(treat==T), F, treat),
censoring = if_else((time > cons_time), 0, 1),
time = pmin(time, cons_time))
fit_model <- coxph(Surv(time, censoring) ~ treat, data = suvival_df)
result_df <- bind_rows(result_df,
data_frame("i" = l,
"Naive" = fit_model$coefficients[1]))
}
結果を確認してみます。
result_longer_df <-
result_df %>%
pivot_longer(cols = -i, names_to = "col", values_to = "val") %>%
mutate(HR = exp(val))
result_longer_df %>%
ggplot() +
aes(x=HR) +
geom_histogram(colour="white", bins = 50) +
geom_vline(aes(xintercept = gamma), colour="red") +
theme_light() +
xlab("HR") +
ggtitle(label = str_c("Histograms of Estimates : True HR = ", round(gamma, 2)))
HR=1と設定したのに、推定された処置群割り当ての影響はそれを下回る=イベント発生が過剰に抑制されている状態にあります。

> result_longer_df$HR %>% summary()
Min. 1st Qu. Median Mean 3rd Qu. Max.
0.3892 0.5059 0.5450 0.5460 0.5819 0.8010
HR=1.5と設定した場合でも同様でした。

> result_longer_df$HR %>% summary()
Min. 1st Qu. Median Mean 3rd Qu. Max.
0.5077 0.6826 0.7290 0.7306 0.7766 1.0048
Immortal time biasへの対応方法
Immortal time biasの対応方法は複数提案されている。
Exclusion Method
処置群において処置が行われるまでの時間を削除して、処置後のみを対象とする方法。
landmerk method
ある時刻(landmerk)までの情報を削除して、処置後のみを対象とする方法。
Mantel-Byar Method
最もImmortal time biasがない推定を行うことができるとされている。
時間依存性共変量を考えた生存時間分析を行う方法。TD-Cox modelとも呼ばれる。
具体的には処置が行われるまでが、一つのサンプルだと考える。つまり処置タイミングまでで打ち切りが行われるサンプルとする。
処置が行われている期間は、左側打ち切りだと考えて2つ目のサンプルとする。
RでImmortal time biasへの対処
Exclusion Method、Landmerk Method、Mantel-Byar Methodでどれほどバイアスが軽減されるかを確認する。
Landmerk Methodにおけるランドマークはt={0.3, 0.5}とした。
gamma_1 = 1.0
base_lambda = 0.6
N = 1000
L = 1000
result_df <- data.frame()
pb <- progress_bar$new(total = L)
for(l in 1:L){
pb$tick()
suvival_df <- data.frame(id = 1:N,
Lambda = base_lambda) %>%
mutate(time1 = rweibull(N, shape=1.0, scale=Lambda),
time2 = rweibull(N, shape=1.0, scale=Lambda*exp(-log(gamma_1))),
treat = rbernoulli(N, p=0.7),
treat_time = rweibull(N, shape=1.0, scale=0.6*Lambda),
cons_time = runif(N, 0, 5)
) %>%
rowwise() %>%
mutate(time = if_else(treat==1, time2, time1),
base_treat = if_else((time2 < treat_time)&(treat==T), F, treat),
censoring = if_else((time > cons_time), 0, 1),
time = pmin(time, cons_time))
suvival_df2 <- tmerge(suvival_df, suvival_df, id = id, censoring = event(time, censoring))
suvival_df2 <- tmerge(suvival_df2, suvival_df2, id = id, treat = tdc(treat_time, base_treat)) %>%
mutate(tstart = if_else(treat == FALSE & !is.na(treat), 0.0, tstart)) %>%
group_by(id) %>%
mutate(group = n()) %>%
filter(!(is.na(treat) & base_treat == FALSE & group==2)) %>%
replace_na(list("treat" = 0))
suvival_df3 <- suvival_df2 %>%
filter(!((time > treat_time) & treat==FALSE)) %>%
mutate(tstop = if_else((time > treat_time), tstop - treat_time, tstop),
surv = Surv(tstop, censoring))
LM_time <- 0.5
suvival_df4_tmp <- suvival_df %>%
filter(time >= LM_time) %>%
mutate(time_event_LM1y = time - LM_time,
time_treat_LM1y = treat_time - LM_time,
flg_treat_LM_1yr = ifelse((treat_time >= LM_time | is.na(treat_time)), 0, 1),
treat_LM1y = ifelse(time_treat_LM1y==1, 0,1)
)
suvival_df4 <- tmerge(suvival_df4_tmp, suvival_df4_tmp, id = id, censoring = event(time, censoring))
suvival_df4 <- tmerge(suvival_df4, suvival_df4, id = id, treat = tdc(treat_time, treat_LM1y)) %>%
replace_na(list("treat" = 0))
LM_time <- 1
suvival_df5_tmp <- suvival_df %>%
filter(time >= LM_time) %>%
mutate(time_event_LM1y = time - LM_time,
time_treat_LM1y = treat_time - LM_time,
flg_treat_LM_1yr = ifelse((treat_time >= LM_time | is.na(treat_time)), 0, 1),
treat_LM1y = ifelse(time_treat_LM1y==1, 0,1)
)
suvival_df5 <- tmerge(suvival_df5_tmp, suvival_df5_tmp, id = id, censoring = event(time, censoring))
suvival_df5 <- tmerge(suvival_df5, suvival_df5, id = id, treat = tdc(treat_time, treat_LM1y)) %>%
replace_na(list("treat" = 0))
fit_nv <- coxph(Surv(time, censoring) ~ base_treat, data = suvival_df)
fit_mb <- coxph(Surv(tstart,tstop, censoring) ~ treat, data = suvival_df2)
fit_em <- coxph(Surv(tstop, censoring) ~ treat, data = suvival_df3)
fit_lm5 <- coxph(Surv(tstop, censoring) ~ treat, data = suvival_df4)
fit_lm10 <- coxph(Surv(tstop, censoring) ~ treat, data = suvival_df5)
result_df <- bind_rows(result_df,
data_frame("i" = l,
"Naive" = fit_nv$coefficients[1],
"Mantel_Byar" = fit_mb$coefficients[1],
"Exclusion" = fit_em$coefficients[1],
"Landmerk5" = fit_lm5$coefficients[1],
"Landmerk10" = fit_lm10$coefficients[1]))
}
生存時間曲線を確認してみる。
result_longer_df <-
result_df %>%
pivot_longer(cols = -i, names_to = "col", values_to = "val") %>%
mutate(val = exp(val))
ggplot(data=result_longer_df, aes(x=val)) +
geom_histogram(colour="white", bins = 50) +
facet_grid(col~.) +
geom_vline(aes(xintercept = gamma_1), colour="red") +
theme_bw() +
ggtitle(label = str_c("Histograms of Estimates : HR = ", round(gamma_1, 2)))

Landmark MethodでもHR=1.0付近に分布しバイアスが小さく、Mantel Byar Methodが最も分散が小さい状態であった。
Landmark Methodはどれぐらいを削除するかを設定しなければならないことも考えると、Mantel Byar Methodを用いるのが良いと言える。
HR=0.7とHR=1.5の場合でも同様であった。


参考文献